(Note: These tutorials are meant to provide
illustrative examples of how to use the AMBER software suite to carry out
simulations that can be run on a simple workstation in a reasonable period of
time. They do not necessarily provide the optimal choice of parameters or
methods for the particular application area.)
Copyright McGee, Miller, and Swails 2009
AMBER ADVANCED TUTORIALS
TUTORIAL 3 - SECTION 3.3
Python Script MMPBSA.py
Dwight McGee, Bill Miller III, and Jason Swails
1)PDBファイルをleap-readyにする。
このシミュレーションでモデル化するシステムは、シグナル伝達カスケードの中心であるヒトH-Rasタンパク質とC-Raf1のラス結合ドメインとの複合体である。下記はRAS-RAF複合体の部分的に平衡化して準備されたPDBファイルである。
この構造には、rasおよびRAFタンパク質と、生理的に必要なGTPヌクレオチドが含まれる。:

tleapによって読まれる変異PDBファイルを準備する必要がある。一貫したprmtopファイルを保証するために、どのシミュレーションを実行する前にも、初期にSection 1で作成されたトポロジーファイルと共に変異体のPDBとトポロジーファイルの作成を強く推薦している。このチュートリアルでは、受容体とリガンドとの間の接合部位にあり、結合エネルギーに顕著な効果を有しているはずの残基イソロイシン21(I21)をアラニン残基21に変異させた。現在のバージョンのコードのみがアラニンへの突然変異をサポートしている。
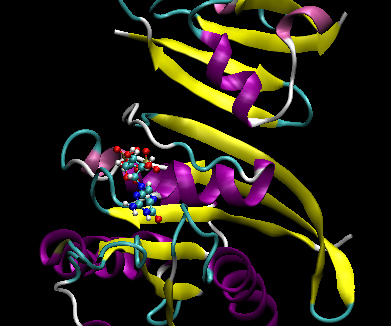
I21は受容体のみで発見されているので、変異体リガンドのPDBファイルを作成する必要はない。したがって、ras-raf.pdb と ras.pdbを変更する必要がある。これを行うには、関与しているアミノ酸の構造についての知知識が必要がある。イソロイシンの側鎖は、-CH(CH3)CH2CH3で、アラニンの側鎖は、-CH3である。イソロイシンの側鎖は、アラニンの側鎖よりも多くの原子を有しているので、原子とPDBファイルから、それに対応する情報(名前、番号、座標等)を除去しなければなら。この変異は、β-炭素(CB)以外のすべての側鎖原子を除去することを意味する。I21では、Ras-RafおよびRasのPDBファイルから原子294から原子305に相当する行を削除しなければならない。システムのために選択した特定のライブラリファイルに基づいてtleapが適切な場所にβ-水素(HB)追加するので、それを自分で追加する必要はない。最後にすべて残基21の原子の「ILE」を「ALA」に変更する。、この手順は、2つの変異体のPDBファイル、ras-raf_mutant.pdb と ras_mutant.pdbを与える。 RAS-RAFのI21A mutationは下図に示す。
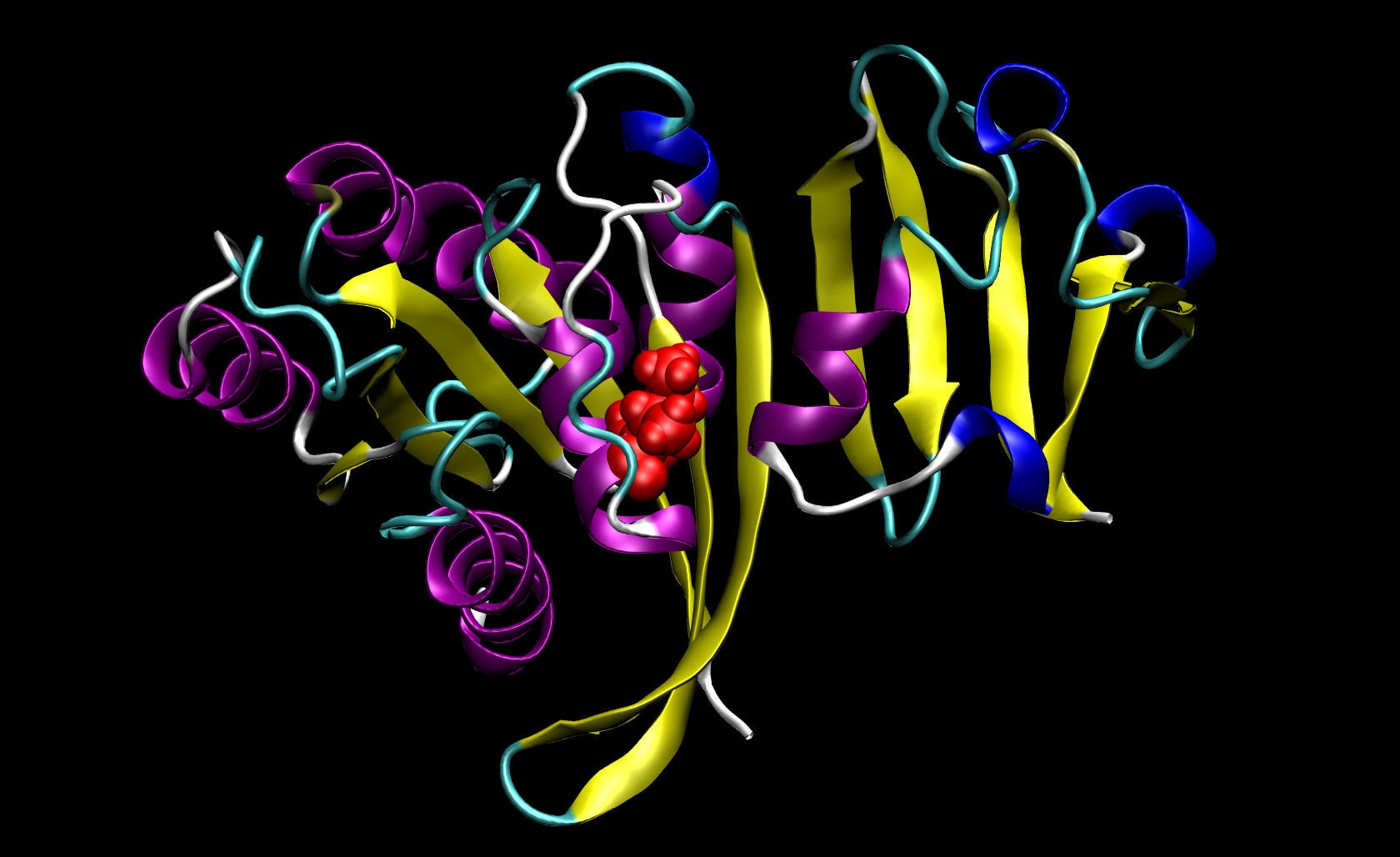
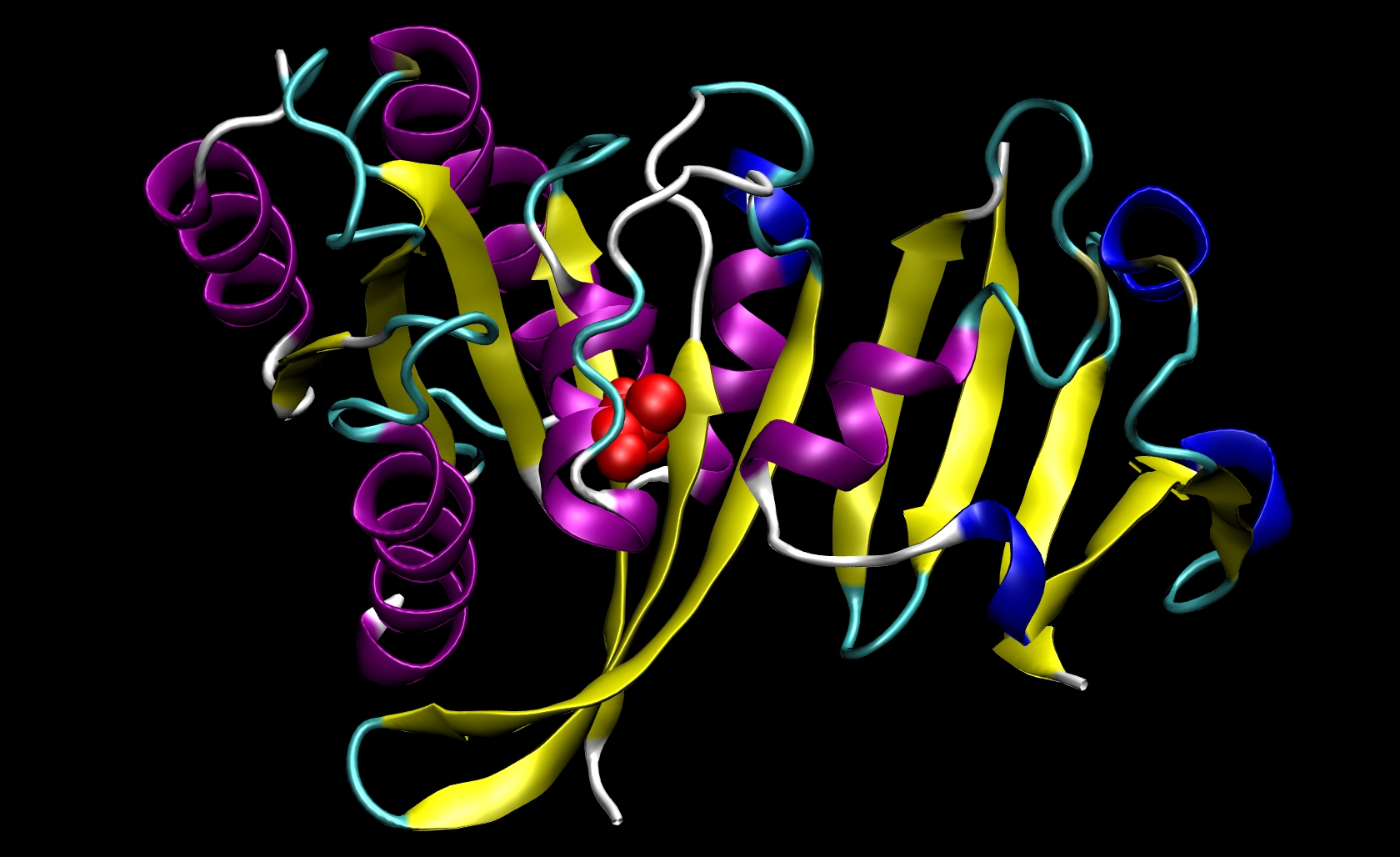
カルボニル炭素(C)以前の原子群以外でCBの後の原子群では、他の変異は同様の手順に従うことができ、PDBファイルから除去することができる。 1つの計算中に実行できる変異は一つのみである。
2)開始トポロジーとcoordinateファイルの構築と、平衡化したシステムを取得するための、シミュレーションを実行。
PDBファイルが作成されたので、tleap使用してその構造に対応するトポロジーとcoordinateファイルを作成する。まず、非突然変異体複合体に対応するファイルを作成する:
> $AMBERHOME/exe/tleap -s -f $AMBERHOME/dat/leap/cmd/leaprc.ff99SB
com = loadpdb ras-raf.pdb
ras = loadpdb ras.pdb
raf = loadpdb raf.pdb
saveamberparm com ras-raf.prmtop ras-raf.inpcrd
saveamberparm ras ras.prmtop ras.inpcrd
saveamberparm raf raf.prmtop raf.inpcrd
また、MDシミュレーションを実行するための溶媒和複合体を作成する必要がある。:
charge com
> Total unperturbed charge: -0.000000
> Total perturbed charge: -0.000000 (Hence there is no need to add counter ions)
solvatebox com TIP3PBOX 12.0
saveamberparm com ras-raf_solvated.prmtop ras-raf_solvated.inpcrd
tleapを終了する前に、作成した変異体のPDBファイルからトポロジーとcoordinateファイルを作成する。:
com_mut = loadpdb ras-raf_mutant.pdb
ras_mut = loadpdb ras_mutant.pdb
saveamberparm com_mut rasraf_mutant.prmtop rasraf_mutant.inpcrd
saveamberparm ras_mut ras_mutant.prmtop ras_mutant.inpcrd
quit
12のファイル(6 .prmtopファイルと6 .inpcrdファイル)が作成された。非突然変異.prmtopと.inpcrdファイルはSection 1 と Section 2で説明されている手順に従って平衡化システムを取得する分子動力学(MD)シミュレーションを実行するために使用される。
MMPBSA.pyを使用して、結合自由エネルギーを計算するための重要なファイルは、非変異トポロジーとcoordinateファイル(ras-raf_alascan.tgz)を使用して実行されたトポロジーファイル(非変異体および突然変異体)とmdcrdファイルである。
3)RAS-Rafの結合自由エネルギーに対してアラニンスキャニング計算を実行
複合体、受容体とリガンドの相互作用エネルギーと溶媒和自由エネルギーを計算し、結合自由エネルギーの推定値を得るために、結果を平均する。mdcrdファイル(複数可)の座標は、「wild-type」構造との比較のために突然変異させた後に続いて同じ計算が変異した構造上で実行する。チュートリアルのこの部分では結合に寄与するエントロピーの計算を実行しないので、厳密には、その結果は真の自由エネルギーを示さないが、同様のシステム間での比較に使用することはできる。Normal Mode Analysis (Nmode)の例としては、Section 3.5にはAMBERのptrajモジュールを使用しQuasi-Harmonicエントロピー計算を実行するために、システムのエントロピーの寄与を計算したり、下記入力ファイルの&general namelistの最後の行をコメントアウトしている。
MM-GBSA法とMM-PBSA法を比較するため両方を使用して結合エネルギー計算をする。MMPBSA.pyでは、以下の入力ファイルを用いて行う。:
| mmpbsa.in |
sample input file for running alanine scanning &general startframe=1, endframe=50, interval=1, verbose=1, / &gb saltcon=0.1 / &pb istrng=0.100 / &alanine_scanning / |
MMPBSA.py用の入力ファイルは、AMBERのsanderモジュールのmdinファイルの設定に同様になるように設計されている。各名前リストは、ampersand (&)で開始し次に名前リストが続く。また、名前リストの終了はbackslash (/)または'&end'で表す。すべての変数の完全なリストは、ユーザーズマニュアルhereを参照する。入力ファイルは、general, pb, と gbとalanine_scanningの4つの名前リストに分割されている。一般的な名前リストは、計算の特定の部分に変数を指定するのではなく、すべての部分に変数を指定するように設計されている。このセットアップでは、受容体をRAS、リガンドはRAFと定義した。'endframe'変数はmdcrdの何フレームで終了するかをセットする。'verbose'変数は計算の終了時にどのファイルを取り除くかを決める。MPIのコマンドの詳細については、マニュアルまたはSection 3.4を参照する。'&gb'と '&pb' ネームリストマーカーは、スクリプトでそれらの名前リスト内で定義され指定された値でMM-GBSAとMM-PBSA計算を実行するかを決めている。「alanine_scanning」名前リストマーカーは、スクリプト内のアラニンスキャニングを初期化する。&alanine_scanning変数群でのみ認識された入力変数は、マニュアルに詳細に記載されている「mutant_only」である。
4つのPythonスクリプト(MMPBSA.py、utils.py、alamdcrd.py、およびinputparse.py)は、インストール時に$AMBERHOME/bin/に置かれている必要がある。スクリプトは、サンダーとpmemdで使用されるものと同様のコマンドラインフラグを使用し(上記の入力ファイルを使用)開始することができる。:
$AMBERHOME/bin/MMPBSA.py -O -i mmpbsa.in -sp ras-raf_solvated.prmtop -cp rasraf.prmtop -rp ras.prmtop -lp raf.prmtop -y *.mdcrd -mc rasraf_mutant.prmtop -mr ras_mutant.prmtop
このコマンドは、対話的にスクリプトを実行し、計算の進捗状況をstdoutとエラーや警告をstderrへ表示する。計算が終わると、計算の各ステップに要した時間を示す。
コマンドライン引数は、シェル認識ワイルドカード(すなわち、*およびbashでは?)で与えることができる。たとえば、コマンドラインで '-y *.mdcrd'は作業ディレクトリ内の '.mdcrd'で終わるすべてのファイルを読み込み、rajectoryとして解析するよう使用することをスクリプトに指示する。
ここで、このスクリプトによって作成されたすべての出力ファイルは: ALASCAN_output.tgz.
スクリプトはptrajを使用して、GBとPBの計算中に分析されたcoordinateである3つの非溶媒和 mdcrdファイル(複合体、受容体、およびリガンド)を作成する。*.mdoutファイルは指定されたすべてのフレームに対するエネルギーを含んでいる。平均構造のPDBファイルには、要求された場合 ptrajによりquasi-harmonicエントロピー計算ための準備するためにすべての一連のスナップショット(RMSを介して)が作成される。MMPBSA.pyによって作成されたすべてのファイルは、最終的な出力ファイルを除き、接頭辞「_MMPBSA_ 'で始まる。 FINAL_RESULTS_MMPBSA.dat
| FINAL_RESULTS_MMPBSA.dat |
| Run on Thu Feb 11 13:11:48 EST 2010 |Input file: |-------------------------------------------------------------- |sample input file for running alanine scanning | &general | startframe=1, endframe=50, interval=1, | verbose=1, |/ |&gb | saltcon=0.1 |/ |&pb | istrng=0.100 |/ |&alanine_scanning |/ |-------------------------------------------------------------- |Solvated complex topology file: ras-raf_solvated.prmtop |Complex topology file: rasraf.prmtop |Receptor topology file: ras.prmtop |Ligand topology file: raf.prmtop |Initial mdcrd(s): bigprod.mdcrd |Mutant complex topology file: rasraf_mutant.prmtop |Mutant receptor topology file: ras_mutant.prmtop |Mutant ligand topology file: raf.prmtop | |Best guess for receptor mask: ":1-166" |Best guess for ligand mask: ":167-242" |Calculations performed using 50 frames. |Poisson Boltzmann calculations performed using internal PBSA solver in sander. | |All units are reported in kcal/mole. ------------------------------------------------------------------------------- ------------------------------------------------------------------------------- GENERALIZED BORN: Complex: Energy Component Average Std. Dev. Std. Err. of Mean ------------------------------------------------------------------------------- VDWAALS -1863.7944 17.1704 2.4283 EEL -17200.7297 75.9366 10.7391 EGB -3142.2247 63.1977 8.9375 ESURF 91.3565 1.3938 0.1971 G gas -19064.5240 77.8536 11.0102 G solv -3050.8682 63.2131 8.9397 TOTAL -22115.3922 51.5332 7.2879 Receptor: Energy Component Average Std. Dev. Std. Err. of Mean ------------------------------------------------------------------------------- VDWAALS -1268.1888 14.2342 2.0130 EEL -11557.0773 71.7127 10.1417 EGB -2444.8629 54.9156 7.7662 ESURF 64.2843 1.1143 0.1576 G gas -12825.2661 73.1118 10.3396 G solv -2380.5786 54.9269 7.7678 TOTAL -15205.8447 36.8422 5.2103 Ligand: Energy Component Average Std. Dev. Std. Err. of Mean ------------------------------------------------------------------------------- VDWAALS -529.3090 9.4198 1.3322 EEL -4684.4720 36.1449 5.1117 EGB -1661.8286 26.5442 3.7539 ESURF 37.0493 0.6185 0.0875 G gas -5213.7811 37.3522 5.2824 G solv -1624.7794 26.5514 3.7549 TOTAL -6838.5604 25.6515 3.6277 Differences (Complex - Receptor - Ligand): Energy Component Average Std. Dev. Std. Err. of Mean ------------------------------------------------------------------------------- VDWAALS -66.2966 4.2751 0.6046 EEL -959.1803 34.9190 4.9383 EGB 964.4668 32.9201 4.6556 ESURF -9.9770 0.3759 0.0532 DELTA G gas -1025.4769 35.1797 4.9752 DELTA G solv 954.4898 32.9223 4.6559 DELTA G binding = -70.9871 +/- 6.6875 0.9457 ------------------------------------------------------------------------------- ------------------------------------------------------------------------------- I21A MUTANT: GENERALIZED BORN: Complex: Energy Component Average Std. Dev. Std. Err. of Mean ------------------------------------------------------------------------------- VDWAALS -1855.4226 17.0765 2.4150 EEL -17210.2882 75.8866 10.7320 EGB -3145.1010 63.2477 8.9446 ESURF 91.8639 1.3913 0.1968 G gas -19065.7108 77.7842 11.0003 G solv -3053.2370 63.2630 8.9467 TOTAL -22118.9478 50.9582 7.2066 Receptor: Energy Component Average Std. Dev. Std. Err. of Mean ------------------------------------------------------------------------------- VDWAALS -1261.9126 14.1817 2.0056 EEL -11566.4419 71.5475 10.1183 EGB -2447.4831 55.0008 7.7783 ESURF 64.5090 1.1105 0.1570 G gas -12828.3545 72.9394 10.3152 G solv -2382.9741 55.0120 7.7799 TOTAL -15211.3287 36.2055 5.1202 Ligand: Energy Component Average Std. Dev. Std. Err. of Mean ------------------------------------------------------------------------------- VDWAALS -529.3090 9.4198 1.3322 EEL -4684.4720 36.1449 5.1117 EGB -1661.8286 26.5442 3.7539 ESURF 37.0493 0.6185 0.0875 G gas -5213.7811 37.3522 5.2824 G solv -1624.7794 26.5514 3.7549 TOTAL -6838.5604 25.6515 3.6277 Differences (Complex - Receptor - Ligand): Energy Component Average Std. Dev. Std. Err. of Mean ------------------------------------------------------------------------------- VDWAALS -64.2010 4.0841 0.5776 EEL -959.3742 34.9114 4.9372 EGB 964.2108 32.9092 4.6541 ESURF -9.6943 0.3800 0.0537 DELTA G gas -1023.5752 35.1495 4.9709 DELTA G solv 954.5164 32.9114 4.6544 DELTA G binding = -69.0588 +/- 6.5302 0.9235 ------------------------------------------------------------------------------- ------------------------------------------------------------------------------- RESULT OF ALANINE SCANNING: (I21A MUTANT:) DELTA DELTA G binding = 1.9283 +/- 9.3470 ------------------------------------------------------------------------------- ------------------------------------------------------------------------------- POISSON BOLTZMANN: Complex: Energy Component Average Std. Dev. Std. Err. of Mean ------------------------------------------------------------------------------- VDWAALS -1863.7944 17.1704 2.4283 EEL -17200.7297 75.9366 10.7391 EPB -3227.2145 64.4523 9.1149 ECAVITY 68.4754 0.7567 0.1070 G gas -19064.5240 6061.1875 857.1813 G solv -3158.7391 64.4568 9.1156 TOTAL -7522.2032 51.2973 7.2545 Receptor: Energy Component Average Std. Dev. Std. Err. of Mean ------------------------------------------------------------------------------- VDWAALS -1268.1888 14.2342 2.0130 EEL -11557.0773 71.7127 10.1417 EPB -2485.3559 54.5638 7.7165 ECAVITY 47.5088 0.4610 0.0652 G gas -12825.2661 5345.3320 755.9441 G solv -2437.8471 54.5658 7.7168 TOTAL -5118.8075 38.9610 5.5099 Ligand: Energy Component Average Std. Dev. Std. Err. of Mean ------------------------------------------------------------------------------- VDWAALS -529.3090 9.4198 1.3322 EEL -4684.4720 36.1449 5.1117 EPB -1684.5802 28.2572 3.9962 ECAVITY 28.1687 0.3939 0.0557 G gas -5213.7811 1395.1865 197.3092 G solv -1656.4114 28.2599 3.9966 TOTAL -2313.4381 24.9082 3.5225 Differences (Complex - Receptor - Ligand): Energy Component Average Std. Dev. Std. Err. of Mean ------------------------------------------------------------------------------- VDWAALS -66.2966 4.2751 0.6046 EEL -959.1803 34.9190 4.9383 EPB 942.7215 33.8861 4.7922 ECAVITY -7.2022 0.3069 0.0434 DELTA G gas -1025.4769 1237.6138 175.0250 DELTA G solv 935.5194 33.8875 4.7924 DELTA G binding = -89.9575 +/- 8.2480 1.1664 ------------------------------------------------------------------------------- ------------------------------------------------------------------------------- I21A MUTANT: POISSON BOLTZMANN: Complex: Energy Component Average Std. Dev. Std. Err. of Mean ------------------------------------------------------------------------------- VDWAALS -1855.4226 17.0765 2.4150 EEL -17210.2882 75.8866 10.7320 EPB -3229.1405 64.8100 9.1655 ECAVITY 68.5521 0.7596 0.1074 G gas -19065.7108 6050.3755 855.6523 G solv -3160.5884 64.8144 9.1661 TOTAL -7520.1586 50.6710 7.1660 Receptor: Energy Component Average Std. Dev. Std. Err. of Mean ------------------------------------------------------------------------------- VDWAALS -1261.9126 14.1817 2.0056 EEL -11566.4419 71.5475 10.1183 EPB -2487.5603 54.6289 7.7257 ECAVITY 47.6466 0.4632 0.0655 G gas -12828.3545 5320.1609 752.3844 G solv -2439.9137 54.6309 7.7260 TOTAL -5118.8820 38.4370 5.4358 Ligand: Energy Component Average Std. Dev. Std. Err. of Mean ------------------------------------------------------------------------------- VDWAALS -529.3090 9.4198 1.3322 EEL -4684.4720 36.1449 5.1117 EPB -1684.5802 28.2572 3.9962 ECAVITY 28.1687 0.3939 0.0557 G gas -5213.7811 1395.1865 197.3092 G solv -1656.4114 28.2599 3.9966 TOTAL -2313.4381 24.9082 3.5225 Differences (Complex - Receptor - Ligand): Energy Component Average Std. Dev. Std. Err. of Mean ------------------------------------------------------------------------------- VDWAALS -64.2010 4.0841 0.5776 EEL -959.3742 34.9114 4.9372 EPB 942.9999 34.0350 4.8133 ECAVITY -7.2632 0.3107 0.0439 DELTA G gas -1023.5752 1235.4872 174.7243 DELTA G solv 935.7367 34.0364 4.8135 DELTA G binding = -87.8385 +/- 8.0665 1.1408 ------------------------------------------------------------------------------- ------------------------------------------------------------------------------- RESULT OF ALANINE SCANNING: (I21A MUTANT:) DELTA DELTA G binding = 2.1190 +/- 11.5368 ------------------------------------------------------------------------------- ------------------------------------------------------------------------------- |
VDWAALS = van der Waals contribution from MM.
EEL = electrostatic energy as calculated by the MM force field.
EPB/EGB = the electrostatic contribution to the solvation free energy calculated by PB or GB respectively.
ECAVITY = nonpolar contribution to the solvation free energy calculated by an empirical model.
DELTA G binding = final estimated binding free energy calculated from the terms above. (kCal/mol)
受容体とリガンドの接合ポテンシャル項の値が、単一のtrajectoryアプローチを使用した複合体接合ポテンシャルを正確にキャンセルする必要があるので、全ガス相エネルギーが報告されていないことに注意する。エネルギーが許容公差内でキャンセルしない場合は、エラーメッセージが発生する。
一般的に複合体形成に有利な静電エネルギーと不利な溶媒和自由エネルギーを予測する。これは、結合粒子の脱溶媒和することと, それらを結合インタフェースに整列させるために使用しなければならないエネルギーを象徴している。
CLICK HERE TO GO TO SECTION 3.4
CLICK HERE TO GO TO MMPBSA.py HOME
CLICK HERE TO GO BACK TO THE INTRODUCTION
(Note: These tutorials are meant to provide
illustrative examples of how to use the AMBER software suite to carry out
simulations that can be run on a simple workstation in a reasonable period of
time. They do not necessarily provide the optimal choice of parameters or
methods for the particular application area.)
Copyright McGee, Miller, and Swails 2009