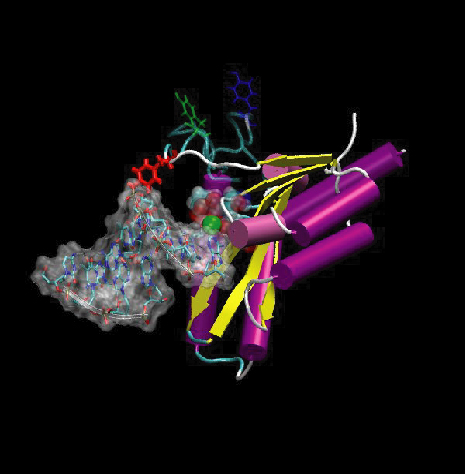

このチュートリアルでは例題として自分の研究プロジェクトの一つを取り上げ、分子動力学シミュレーションをAmberにより実行する基本的ステップを案内する。これからシミュレーションに使う分子は HIV-1 integrase のコアドメインです。しかし、作業に入る前にこの分子に関する背景を記します。 このインテグラーゼはHIV−1ウイルスの複製に必要な3酵素の一つで、その基本的機能はウイルスDNAをゲノムに挿入することです。 全長288残基のタンパク質ですが、非常に溶媒に溶け難くこのインテグラーゼの全長構造を実験的に得ることは困難であるとされている。幸い、タンパク質分解による分析で本酵素は3つの独立したフォールディングドメインに分けられることが示されている: the N-terminal domain、 the C-terminal domainおよび the catalytic core domain です。 触媒ドメインの高解像度結晶構造を含め、 実験的に決定されたそれぞれのドメイン構造は報告されている。これらの結晶構造から、保存された活性中心近くに構造を持たない表面ループが存在していることが判明している。タンパク質の表面ループは触媒活性に関与していることが多いので、ループの構造動力学の理解はタンパク質の触媒機構を理解する上で重要である。本インテグラーゼの場合、突然変異導入研究はインテグラーゼの活性に著しい影響を持つ2、3の残基の存在を明らかにした。表紙の図は10ns分子動力学trajectoryの結果を示している。インテグラーゼのコアドメインはcatoon表示してあり、DNA基質は表面表示で示してある。表面ループの上に、保存残基であるTyr143が青 で強調してあり、緑、赤で異なったステージでのループの構造変化を示している。結合サイトにドッキングされたDNA基質は表面表示で表示してある。
これらの情報をAMBERによりいかにして得るかを次ぎに記述する。
[next]