Reference Material and Information Sources
-
The Amber Website (http://www.ambermd.org/)
-
The Amber Mailing List Archive (http://archive.ambermd.org/)

ここではamber tutorialの日本語訳を掲載します。本来のtutorialはbasic、advanced、analysis_specificに分けてあるけれど全部のtutorialを訳す予定は無いので当面は一カ所に載せます。私が必要としたものだけを載せますから後は別の所を探してください。
(Note:このチュートリアルはamberのソフトをシミュレーションに使うのに適当な時間内で簡単なパソコンで実行出来る例を提供する。従って、それらは特殊なアプリケーション分野での最適のパラメーターや方法を提供するものではない。)
The Amber Website (http://www.ambermd.org/)
The Amber Mailing List Archive (http://archive.ambermd.org/)

 AntechamberはAmberとセットで送付されるツールセットで、有機分子の"prepを作成しLEaPで読み込んでprmtopとinpcrdファイルを作れるようにする。Antechamber suiteは"general AMBER force field
(GAFF)"とともに使用するようにデザインされており、有機医薬化合物やその他の有機分子を含むシミュレーションに理想的である。このチュートリアルではBMS's HIVの逆転写酵素阻害剤sustiva (efavirenz)のleap inputファイルを作成するためにいantechamberを用いる。その後 HIV-RTに結合したsustivaのシミュレーションをセットアップする。 (Ukrainian translation)
AntechamberはAmberとセットで送付されるツールセットで、有機分子の"prepを作成しLEaPで読み込んでprmtopとinpcrdファイルを作れるようにする。Antechamber suiteは"general AMBER force field
(GAFF)"とともに使用するようにデザインされており、有機医薬化合物やその他の有機分子を含むシミュレーションに理想的である。このチュートリアルではBMS's HIVの逆転写酵素阻害剤sustiva (efavirenz)のleap inputファイルを作成するためにいantechamberを用いる。その後 HIV-RTに結合したsustivaのシミュレーションをセットアップする。 (Ukrainian translation)
以下の二つのチュートリアルは MD simulationの結果をCPPTRAJで分析することに焦点をあてている。
TUTORIAL C0: An Introduction to CPPTRAJ
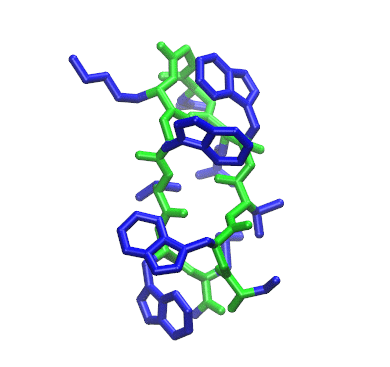 このチュートリアルはCPPTRAJを用いてtrajectory分析をするための基本的なイントロダクションを示している。従って topologies と trajectoriesをロードし, dataを処理し, さらにdata setsを使うためにCPPTRAJをinteractively もしくは batchmodeで作業することを解説している。
このチュートリアルはCPPTRAJを用いてtrajectory分析をするための基本的なイントロダクションを示している。従って topologies と trajectoriesをロードし, dataを処理し, さらにdata setsを使うためにCPPTRAJをinteractively もしくは batchmodeで作業することを解説している。
By Daniel R. Roe
TUTORIAL C1: RMSD Analysis in CPPTRAJ
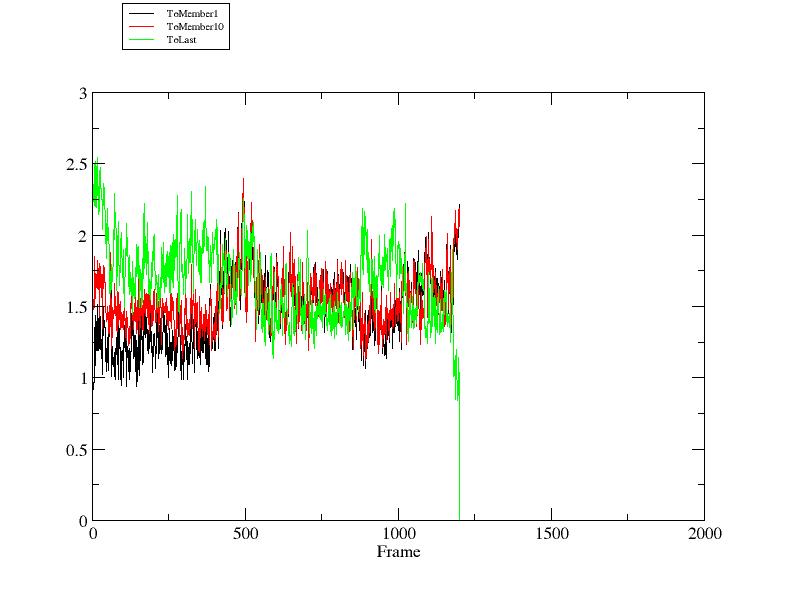 このチュートリアルはCPPTRAJを用いてtRMSD計算をするための基本的なイントロダクションを示している。従ってreference structuresをロードし、異なるtopologiesを持つreferencesに対するRMSDを計算することもカバーしている。
このチュートリアルはCPPTRAJを用いてtRMSD計算をするための基本的なイントロダクションを示している。従ってreference structuresをロードし、異なるtopologiesを持つreferencesに対するRMSDを計算することもカバーしている。
By Daniel R. Roe